DrugFinder_Druggable_Protein_Identification_Model_Based_on_Pre_Trained
前面的话
摘要,随着近些年来医学领域的发展,对于药物蛋白的需求越来越大,使用常规的根据结构识别蛋白的方法费时费力,因此,越来越多的研究者开始使用序列化的方式来识别药物蛋白.
补充知识
1
2
3
4
5
6
7
8
9
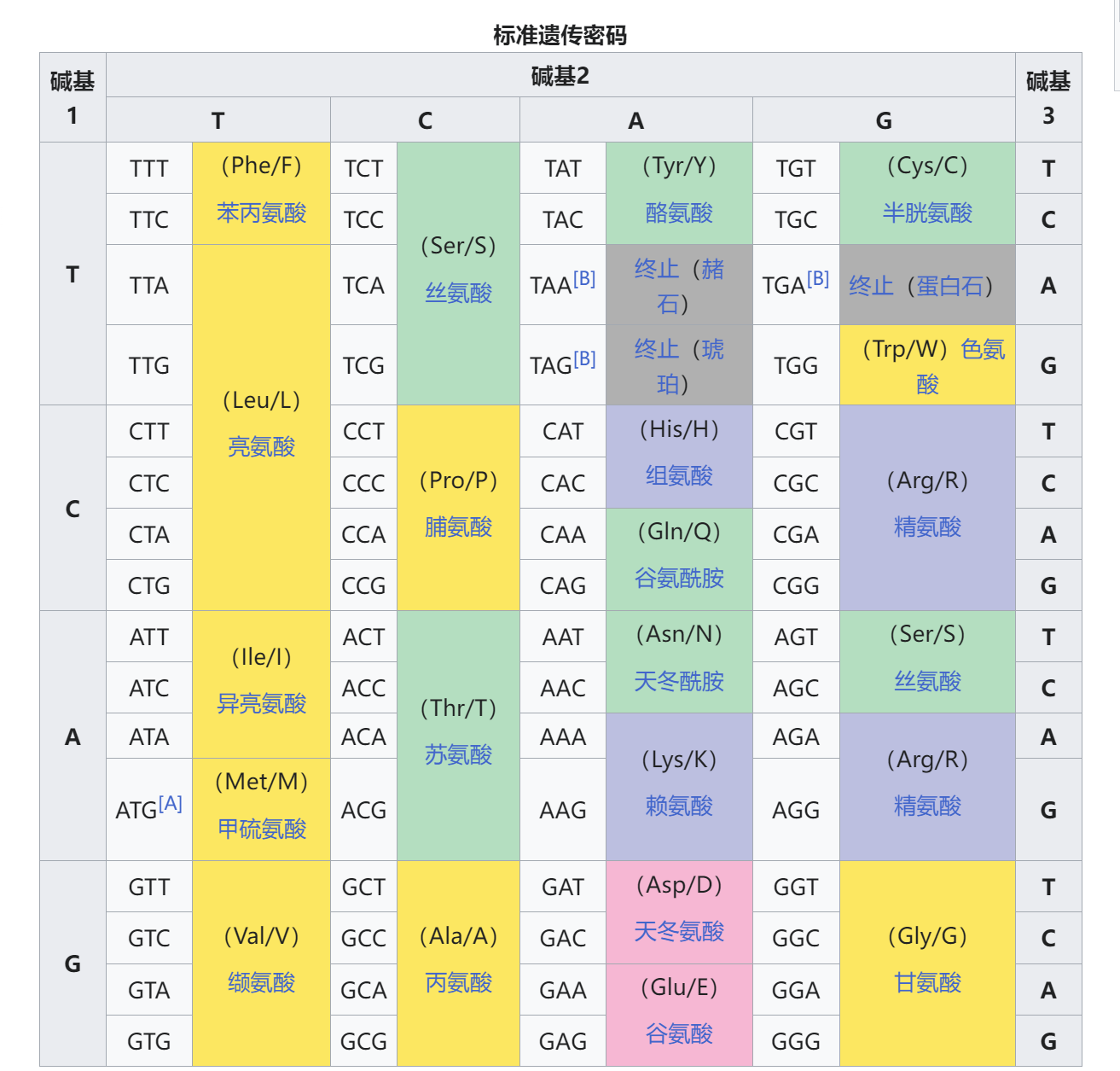
碱基有5种ATCGU
密码子有64种,映射到21种氨基酸上
蛋白氨基酸主要有二十一种,包括甘氨酸、丙氨酸、缬氨酸、亮氨酸、异亮氨酸、甲硫氨酸(蛋氨酸)、脯氨酸、色氨酸、丝氨酸、酪氨酸、半胱氨酸、苯丙氨酸、天冬酰胺、谷氨酰胺、苏氨酸、天门冬氨酸、谷氨酸、精氨酸、组氨酸,硒半胱氨酸和赖氨酸,前十九种较为常见,也称为“二十种氨基酸”,最后两种分别用通常的终止密码子UGA和UAG编码,在少数蛋白质中出现。
正因为氨基酸的种类少,所以可以将其抽象成数字来替代,之后再用一些人工智能的算法可以去提取一些特征,比如用CNN或者SVM,有了一段氨基酸序列,实际就可以将其转化成对应的蛋白质了
但是这种方式真的好吗,蛋白质复杂多变,不仅仅在于其氨基酸序列的变化,实际上相同序列的蛋白质因为其空间折叠的方式不同也有可能是不一样的,然后这种不同的蛋白质可能会导致千差万别的结果,比如一个有毒,一个没毒(类似青霉胺的手性,D-青霉胺常用于螯合疗法及治疗类风湿性关节炎,L-青霉胺则因为会抑制吡哆醇(一种人体必需的维他命B)在人体的作用而具有毒性。) 这个还有待商榷 :[
为此,作者提出了一种序列化的方式来识别/分类药物蛋白,并将其命名为DrugFinder(名字其实起的有点俗了)的模型.使用了前人的预训练模型Prot_T5_Xl_Uniref50
| 氨基酸(中文名) | 氨基酸(英文名) | 单字母代码 |
|---|---|---|
| 丙氨酸 | Alanine | A |
| 精氨酸 | Arginine | R |
| 天冬酰胺 | Asparagine | N |
| 天冬氨酸 | Aspartic acid | D |
| 半胱氨酸 | Cysteine | C |
| 谷氨酸 | Glutamic acid | E |
| 谷氨酰胺 | Glutamine | Q |
| 甘氨酸 | Glycine | G |
| 组氨酸 | Histidine | H |
| 异亮氨酸 | Isoleucine | I |
| 亮氨酸 | Leucine | L |
| 赖氨酸 | Lysine | K |
| 甲硫氨酸 | Methionine | M |
| 苯丙氨酸 | Phenylalanine | F |
| 脯氨酸 | Proline | P |
| 丝氨酸 | Serine | S |
| 苏氨酸 | Threonine | T |
| 色氨酸 | Tryptophan | W |
| 酪氨酸 | Tyrosine | Y |
| 缬氨酸 | Valine | V |
流程
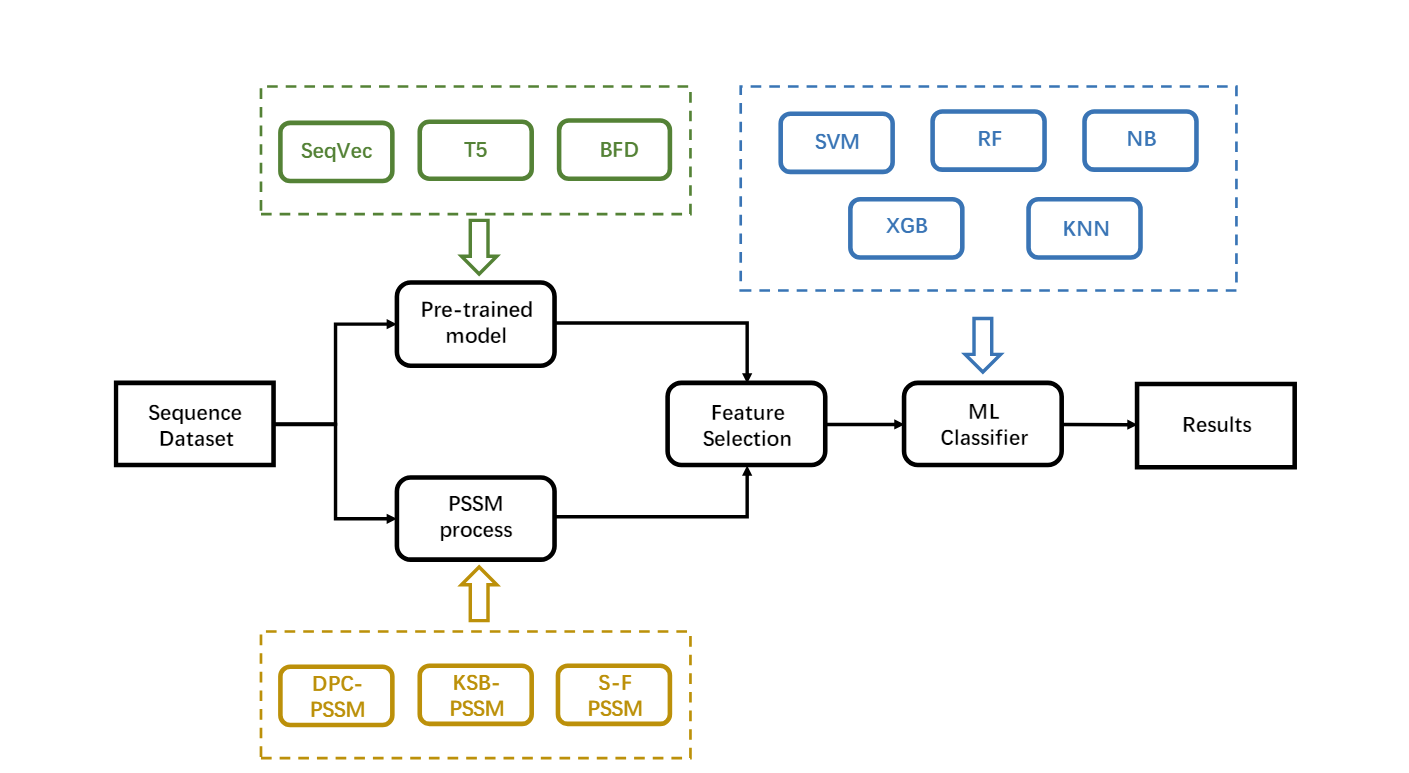
1.特征提取:
将蛋白质序列输入预训练模型 (Prot_T5_Xl_Uniref50 (T5), Prot_Bert_BFD (BFD), SeqVec) 生成嵌入向量 (1024 维)。
使用 Blast 软件包捕获蛋白质序列的 PSSM 信息,并将其处理成 KSB-PSSM (400 维)、DPC-PSSM (400 维) 和 S-FPSSM (400 维) 向量。
1
2
BLAST全称Basic Local Alignment Search Tool ,即基于局部序列比对算法的搜索工具。 是由美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)开发和管理的一套生物大分子一级结构序列比对程序。
2.特征选择:
使用随机森林 (RF) 对上述提取的特征进行选择,去除冗余特征并提高模型性能。
分类:
使用机器学习分类器 (SVM, RF, NB, XGB, KNN) 对选择后的特征进行训练和测试。
算法细节
- 预训练模型: 本文使用了三种基于 Transformer 架构的预训练模型:
- ProtT5-XL-UniRef50 (T5): 基于 Google 的 T5 模型,在 UniRef50 数据库上进行训练。
- ProtBert-BFD: 基于 Google 的 BERT 模型,在 Big Fantastic Database (BFD) 上进行训练。
- SeqVec: 基于 ELMo 模型,在 UniRef50 数据库上进行训练。
- 实现方式:
- 将蛋白质序列输入预训练模型,获取模型最后一层的隐藏状态 (hidden states)。
将每个氨基酸对应的隐藏状态取平均值,得到固定长度的蛋白质序列嵌入向量 (1024 维)。
- PSSM 计算: 使用 Blast 软件的 PSI-BLAST 程序搜索蛋白质序列数据库 (如 UniRef50),生成 PSSM 矩阵。PSSM 矩阵的每一行代表蛋白质序列中的一个氨基酸,每一列代表一种氨基酸类型,矩阵元素表示该位点突变成该氨基酸类型的对数似然比。
- PSSM 特征: 本文使用了三种基于 PSSM 的特征:
- DPC-PSSM (Dipeptide Composition): 计算蛋白质序列中每对相邻氨基酸的出现频率,并结合 PSSM 信息,生成 800 维特征向量。
- 公式:
Di,j = (1/(L-1)) * Σ(Dk,i * Dk+1,j)其中
Dk,i表示第 k 个氨基酸是第 i 种类型的概率 (从 PSSM 中获取)。 - KSB-PSSM (K-Separated Bigrams): 计算蛋白质序列中距离为 k 的氨基酸对的出现频率,并结合 PSSM 信息,生成 400 维特征向量。
- 公式:
Ki,j(k) = (1/(L-k)) * Σ(Kt,i * Kt+k,j) - S-FPSSM (Secondary-FPSSM): 对 PSSM 矩阵进行行变换,生成 400 维特征向量。
- 公式:
S(i)j = Σ(fpk,j * δk,i)其中
fpk,j表示 FPSSM 矩阵中第 k 行第 j 列的元素 (FPSSM 是 PSSM 的过滤版本),δk,i是一个指示函数。 - 算法: 随机森林 (Random Forest)
- DPC-PSSM (Dipeptide Composition): 计算蛋白质序列中每对相邻氨基酸的出现频率,并结合 PSSM 信息,生成 800 维特征向量。
- 实现方式:
- 使用全部特征 (1024 维 embedding + 1200 维 PSSM 特征) 训练随机森林分类器。
- 根据特征在随机森林模型中的重要性进行排序。
- 选择重要性排名靠前的特征,构建新的特征子集。
- 特征子集大小: 本文尝试了不同的特征子集大小 (100, 300, 500, … , 2000),并通过实验确定了最佳的特征维度。
1
2
3
4
5
6
* 分类器:
* 支持向量机 (SVM)
* 随机森林 (RF)
* 朴素贝叶斯 (NB)
* 极端梯度提升 (XGBoost)
* K 近邻 (KNN)
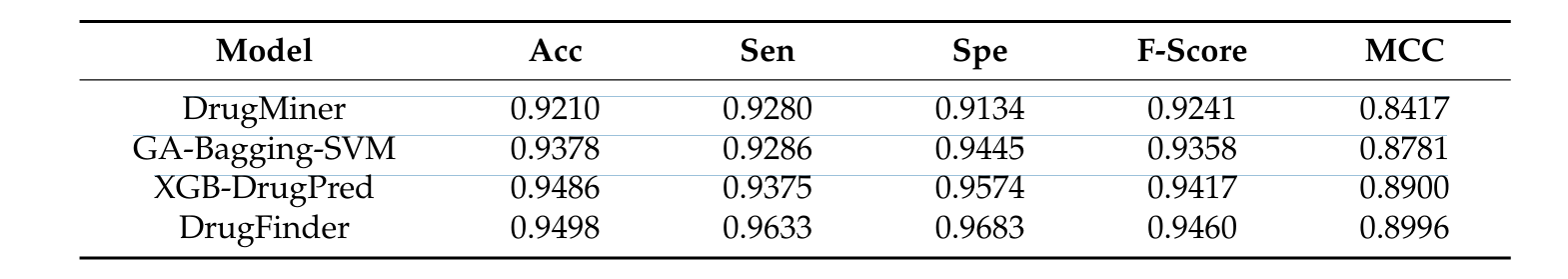
结果
bad news
学长有把论文代码放在GitHub上面
实际git clone下载下来后,配了两个小时环境还没配出来
这个bio-embeddings太逆天了
也不知道学长机器用的啥环境,估计是Linux,否则用不到llvm和clang操作
也不知道有生之年能不能成功复现:-(
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
2 warnings generated.
Compiler msvc
building 'blis.cy' extension
creating build\temp.win-amd64-cpython-310
creating build\temp.win-amd64-cpython-310\Release
creating build\temp.win-amd64-cpython-310\Release\blis
D:\Programs\LLVM\bin\clang.exe -IC:\Users\minec\AppData\Local\Temp\pip-install-m0wu15nh\blis_d8117c58ee0d4c58944c9b84bde1b10e\include -IC:\Users\minec\AppData\Local\Temp\pip-install-m0wu15nh\blis_d8117c58ee0d4c58944c9b84bde1b10e\blis\_src\include\linux-x86_64 -ID:\Programs\Anaconda\envs\drug\include -ID:\Programs\Anaconda\envs\drug\Include -c blis\cy.c -o build\temp.win-amd64-cpython-310\Release\blis\cy.o -std=c99
In file included from blis\cy.c:4:
In file included from D:\Programs\Anaconda\envs\drug\include\Python.h:130:
D:\Programs\Anaconda\envs\drug\include\cpython/pytime.h:120:59: warning: declaration of 'struct timeval' will not be visible outside of this function [-Wvisibility]
120 | PyAPI_FUNC(int) _PyTime_FromTimeval(_PyTime_t *tp, struct timeval *tv);
| ^
D:\Programs\Anaconda\envs\drug\include\cpython/pytime.h:127:12: warning: declaration of 'struct timeval' will not be visible outside of this function [-Wvisibility]
127 | struct timeval *tv,
| ^
D:\Programs\Anaconda\envs\drug\include\cpython/pytime.h:132:12: warning: declaration of 'struct timeval' will not be visible outside of this function [-Wvisibility]
132 | struct timeval *tv,
| ^
blis\cy.c:20396:5: error: expression is not assignable
20396 | ++Py_REFCNT(o);
| ^ ~~~~~~~~~~~~
blis\cy.c:20398:5: error: expression is not assignable
20398 | --Py_REFCNT(o);
| ^ ~~~~~~~~~~~~
blis\cy.c:20685:5: error: expression is not assignable
20685 | ++Py_REFCNT(o);
| ^ ~~~~~~~~~~~~
blis\cy.c:20687:5: error: expression is not assignable
20687 | --Py_REFCNT(o);
| ^ ~~~~~~~~~~~~
blis\cy.c:20924:5: error: expression is not assignable
20924 | ++Py_REFCNT(o);
| ^ ~~~~~~~~~~~~
blis\cy.c:20926:5: error: expression is not assignable
20926 | --Py_REFCNT(o);
| ^ ~~~~~~~~~~~~
blis\cy.c:21738:18: error: no member named 'tp_print' in 'struct _typeobject'
21738 | __Pyx_EnumMeta.tp_print = 0;
| ~~~~~~~~~~~~~~ ^